

RNA-蛋白质相互作用在转录后基因调控中至关重要。随着RNA测序技术和高效液相质谱联用的蛋白质组学技术的快速发展,将这些创新的化学生物工具与生物学研究相结合,预示了在细胞水平研究分子的互作将成为可能。
转录后基因调控在生物过程中起着十分重要的作用。很大程度上,潜在的分子机制是由RNA转录本与大量RNA结合蛋白(RNA binding proteins, RBPs)在物理层面的互作介导的,这些蛋白调节RNA剪接(splicing)、稳定性(stability)、核转运(nuclear export)和翻译(translation)等特性。因此,阐释这些RBPs的RNA结合偏好并绘制RBPs蛋白质组图有助于揭示RNA-蛋白质结合互作的生物学功能。
另外,最新研究表明RNA修饰作为RNA-蛋白质复合物的调节元件,增加了互作的复杂性。尽管RNA-蛋白质相互作用的早期研究仅限于单个蛋白复合物的研究,但是RNA测序和蛋白质组学在技术层面的进步使得研究人员能够从转录组和蛋白质组两个维度进行分析。
值得注意的是,在完整的生物样品如细胞和动物组织中进行UV介导的蛋白质-RNA复合物光交联,在开发以高通量测序研究RBP-RNA互作的方法中发挥了重要作用。此外,化学生物学方法如用人工核苷酸进行代谢标记,生物正交化学(bio-orthogonal chemistry)和蛋白质工程学,也都应用到阐明蛋白质-RNA互作组学数据研究中。本文作者将重点介绍细胞中RNA-蛋白质互作的高通量组学技术最新进展,并展望该领域未来的研究发展方向。
研究RNA-蛋白结合位点印记的方法
Approaches to footprint RNA-protein binding sites
理解RBP生物学功能和分子机制的关键是鉴定所有RNA转录本的集合。此外,RBP的RNA结合位点或“足迹”(footprint)也可以帮助我们进一步了解其生物学活性。因此早期的研究主要依靠体外选择,利用RBP对固定序列结合的偏好性在随机RNA库中寻找特定规律。这种方法能够鉴定出某种或某类RBP对哪种规律序列有结合偏好性,这种高亲和力的结合序列也称为motif,此方法可以在转录组测序中挖掘出相关的RNA序列。体外选择方法操作简单易行,然而并不能鉴定到生理条件下真实RNA-蛋白互作事件,因此只能限定在对从活体样本中分离的RNA-蛋白复合物的鉴定应用上。
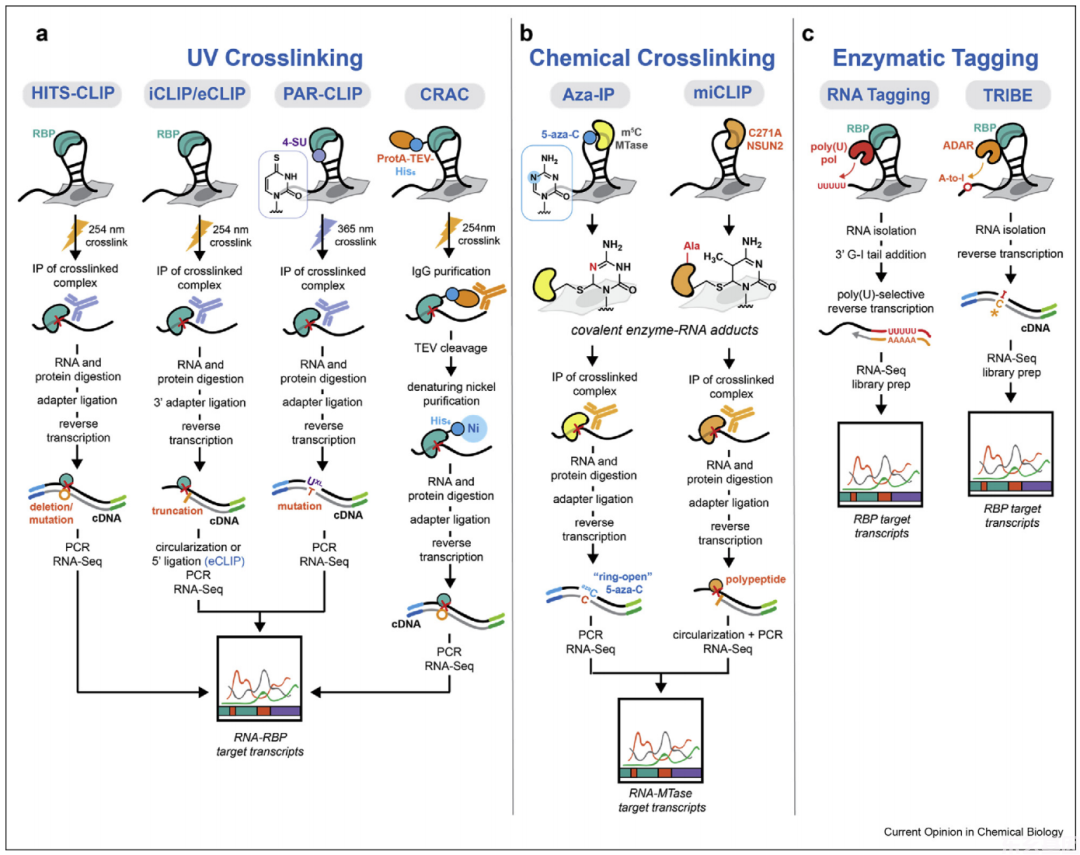
图1. 三类主流的RNA-蛋白结合位点高通量测序鉴定方法
Darnell等人在2008年发表了公认的第一篇RNA-蛋白互作高通量测序方法论文(Pubmed ID:18978773),这种方法被称为HITS-CLIP或CLIP-seq。HITS-CLIP结合了几种关键方法,通过高交联和免疫沉淀分离天然RNA-蛋白质复合物。该方法的核心是紫外线诱导的光交联,该交联可以在完整细胞上操作,并且交联的RNA-蛋白质互作效率比蛋白质-蛋白质的效率高很多。RNA-蛋白质发生交联后,就可以在严格的免疫沉淀条件下分离出低亲和度的蛋白质-RNA互作,并通过逆转录cDNA和测序来鉴定相关的RNA。
通常在免疫沉淀之前和之后对RNA进行部分酶消化,以更精确地定位RBP足迹。HITS-CLIP是许多基于UV-光交联的CLIP方法的先驱,这些方法的目的是为了绘制单个RBP在转录组范围内的RNA结合图谱。图1a还罗列了包括CRAC、PAR-CLIP、iCLIP和eCLIP以及对原有HITS-CLIP进行改进的方法。这些改进通常分为以下几类:
(1)利用4-硫尿苷(4-SU)或6-硫鸟嘌呤(6-SG)对细胞RNA进行代谢标记从而增强光交联作用;
(2)优化免疫沉淀和分离条件;
(3)改进cDNA文库建库方法和生物信息分析算法,旨在绘制具有单核苷酸分辨率的RBP结合位点信息(通常依赖于交联诱导突变的鉴定)。
当前,这些改进的CLIP方法代表了在整个转录组中绘制RBP足迹的最新技术,并已被广泛应用。尽管UV交联是可以应用于几乎所有RBP研究,但是RNAm5C甲基转移酶的独特催化机制使其能够通过化学手段与底物RNA交联。图1b报道了两种交联方法来绘制m5C甲基转移酶底物,即Aza-IP和miCLIP。在Aza-IP方法中,RNA-蛋白质交联是由5-氮杂胞苷(5-azacytidine)介导的,通过代谢标记在转录组范围内整合。而在miCLIP方法中,通过使用突变甲基转移酶与其底物胞苷残基结合交联后,运用类似UV交联的CLIP流程方法进行后续的免疫沉淀和RNA测序分析。
非交联策略也已应用于RBP结合RNA图谱相关研究中。图1c中的TRIBE原理是当腺苷脱氨酶(ADARs)接近RNA转录本时,能够催化腺苷对肌苷(A:I)编辑(例如通过与感兴趣的RBP融合)。此方法不需要纯化RNA-蛋白复合物,因为在逆转录过程中肌苷主要转化为C,并且通过测序和生物信息学分析可以直接确定它的存在。图1c中使用更活跃的ADAR突变体的方法叫HyperTRIBE,是TRIBE的增强版。Wickens等人开发了另一种非交联的方法,名为“RNA标签”(RNA tagging),主要依赖线虫的聚U聚合酶(PUP-2)与感兴趣的RBP融合,导致polyU尾巴沉积在RBP相关的RNA上,然后在cDNA文库生成过程中选择性富集PolyU标签。TRIBE和RNA tagging的主要优点是不需要纯化RNA-蛋白质复合物,但是它们都涉及将酶与目标RBP融合,这可能会影响RNA结合效率,且无法提供单碱基分辨率水平的结合数据。
RNA结合蛋白的蛋白质组分析方法
Approaches to profile the RNA-binding proteome
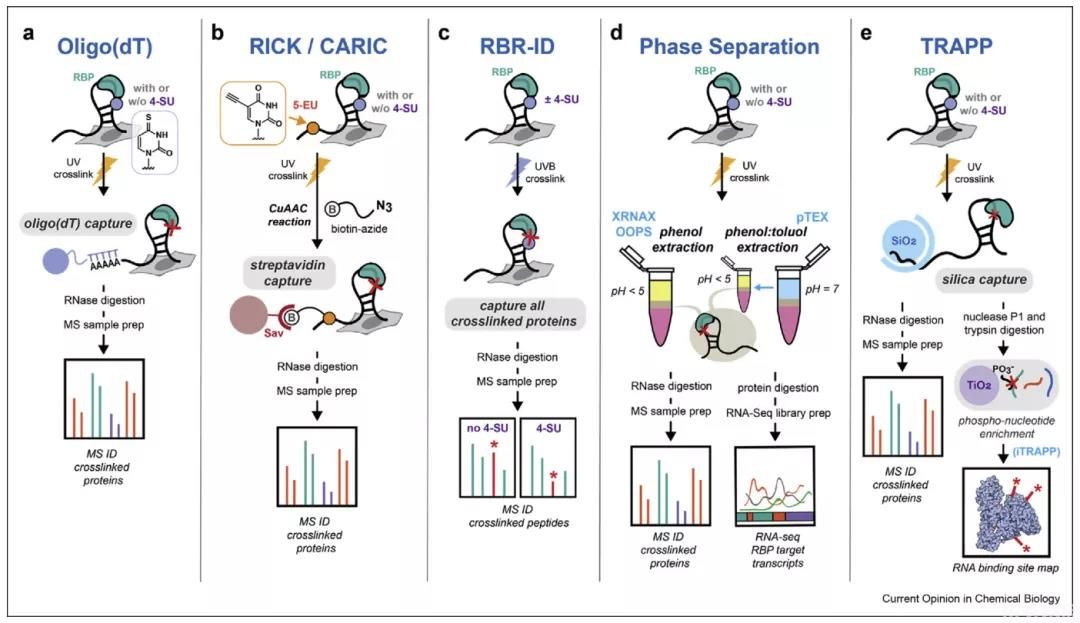
图2. RNA结合蛋白检测方法
CLIP的测序方法可以提供单个RBP结合到RNA的信息,但无法研究RNA结合蛋白对应的蛋白质组信息。若要发现新的RBP及其在蛋白质组范围内的特征,有必要将蛋白质组学应用于RBP分析。由于蛋白通量较低且无法扩增,使得蛋白质组学研究比RNA测序更具有挑战性,在最近十年,出现了多种用于细胞RBP互补蛋白质组学特征研究的方法,这些方法都是建立在最初CLIP实验开发的RNA-蛋白质交联方法的基础上。
Hentze和Landthaler实验室独立开发了第一种从细胞中分离RBP的技术,称为“交互作用捕获”(interactome capture)(图2a)。在其各自的方法中,首先将RBP通过UV交联到RNA上,然后通过与oligo-dT珠杂交从细胞中富集polyA RNA-蛋白复合物,在酶消化RNA后,运用质谱的蛋白质组学来鉴定分离的RBP。由于核酸杂交耐受高盐和离子去污剂,因此可以在严格的变性条件下进行oligo-dT富集,从而从细胞里游离蛋白中分离出RNA交联蛋白。互作捕获法已被广泛用于研究RBP与mRNA的互作,可用于254nm UV交联或更长波长的辐射结合4-SU标记的RNA。已知在HeLa和HEK293细胞中鉴定出约800个RBP,其中许多是首次发现的RBP。
非依赖ployA RNA的另一种方法是RICK/ CARIC法,可用于分析与RNA转录本相关的RBP,而不论其是否有polyA(图2b)。这种方法依靠RNA上的5-乙炔基尿苷(5-EU)标记和生物素标记来富集与新生转录本(nascent transcripts)交联的RBP,并为oligo-dT的富集提供结果。研究RBP要有所突破就必须考虑在开发方法上有更大的通用性,不能只局限于分析与polyA化RNA相关的RBP,需要开发能够更深入了解与RNA结合有关的蛋白质结构域和氨基酸残基的方法。
Bonasio等人开发了一种名为RBR-ID的方法(图2c),通过分析交联的胰蛋白酶肽来鉴定RBP结合区。RBR-ID并不是测量特定的交联肽-寡核苷酸种类,而是利用了亲本(非交联)胰蛋白酶肽的信号强度随之降低的优势。在胚胎干细胞的核蛋白质组上使用这种检测方法,可以在肽水平上鉴定出约800个RBP结合区,包括染色质调节剂中几个以前未知的RNA结合域。此外,RBR-ID法不需要对RNA进行富集,因此可以提供RNA结合蛋白质组的无差别快照信息(unbiased snapshot)。
RNA修饰相关蛋白研究方法进展
Approaches to study RNA modification–associated proteins
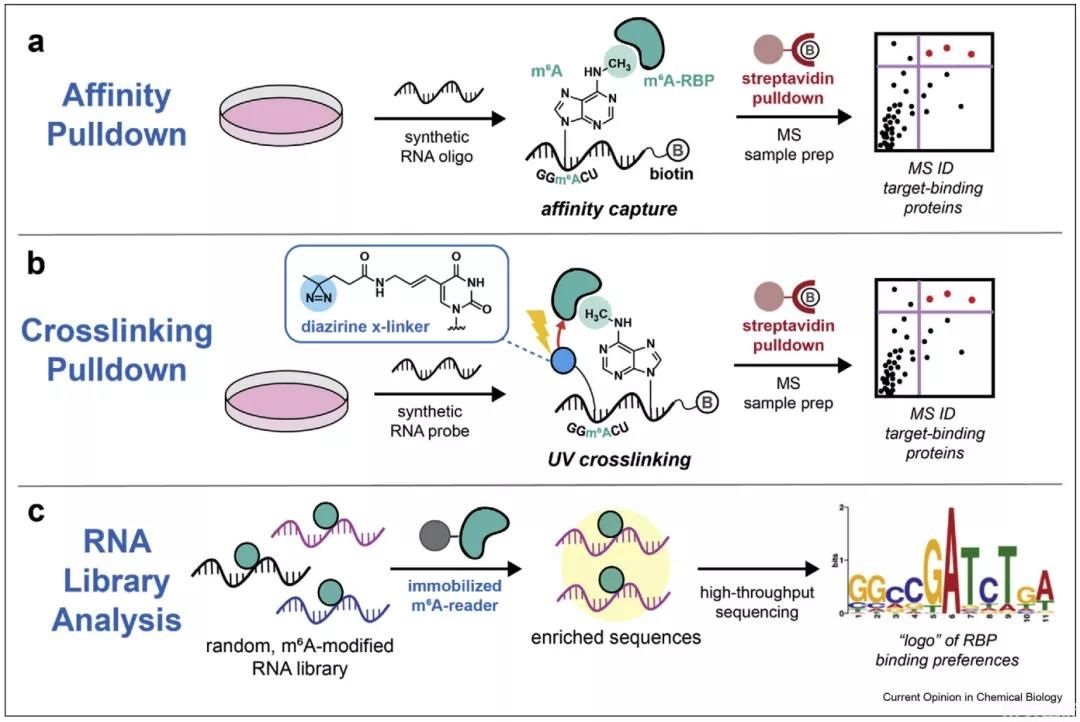
图3. 研究依赖RNA修饰的RBP的方法
由于RNA上的修饰水平较低,因此CLIP或RNA互作捕获等方法不太适用于研究与RNA修饰相关的RBP(或者叫阅读蛋白reader)。同时,作者也指出现在仍然缺乏在细胞修饰位点附近进行偏向光交联(bias photocrosslinking)的方法。因此鉴定这些RNA修饰阅读蛋白很大程度上依赖于合成一段带有m6A修饰motif序列的oligo寡核苷酸碱基序列,这段oligo上带有生物素等标记,方便进行pulldown。该方法已经成功鉴定了多种新型的m6A阅读蛋白,如陈建军课题组的黄慧琳博士(现任职于中山大学肿瘤防治中心)发表在2018年的Nature Cell Biology上的利用pulldown技术成功鉴定了IGF2BP家族新型的m6A阅读蛋白的研究。
新的研究方法有助于扩大结合RNA的蛋白组学
经典的RBPs一般包含RNA结合区域,但是最近的研究进展表明,新发现的RBPs并不包含已知的RNA结合区域。因此,如果只依靠蛋白序列和结构信息,是无法准确预测该蛋白是一种RBP。当利用UV交联法促进RNA与蛋白之间形成共价键,接着Oligo(dT)捕获poly(A)尾的蛋白,用于蛋白组分析以鉴定RBPs。但是这种方法局限在于只能鉴定结合含有poly(A)尾RNA的蛋白。后来的研究发展了一种click-chemistry-assisted RNA interactome capture (CARIC)研究方法,利用炔基尿苷类似物代谢性标记RNAs,可以捕获任意RNAs,而不局限于含有poly(A)尾的RNA。结合以上两种研究方法,大量的RBPs逐渐被发现,提示大约5%的人蛋白组属于RBPs。
以感兴趣的RNA为中心,寻找与该RNA相互作用的蛋白质,分为以下三种研究方法:
1. 体外RNA-centric法
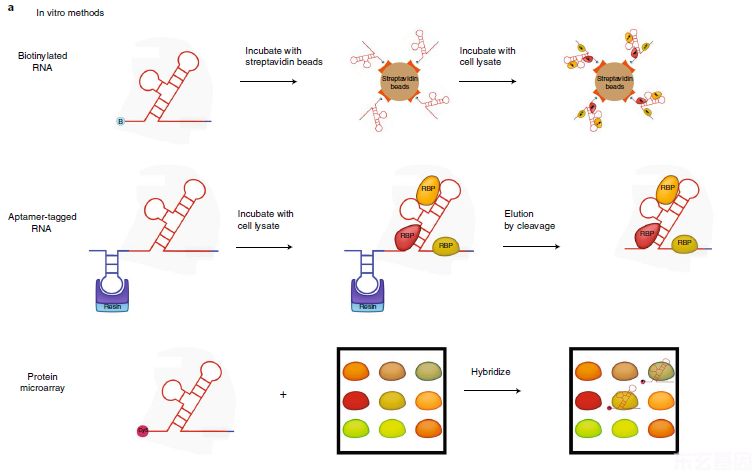
图1 体外方法:顶部图, 合成5'或3'末端标记生物素的RNA并与链霉亲和素结合。
重组蛋白或细胞提取蛋白与RNA结合。洗涤后,将珠子煮沸以洗脱并鉴定与RNA结合的蛋白质;中间图表示适体(S1,Cys4)标记的RNA捕获方法。感兴趣的RNA是体外转录合成的带有RNA标签(蓝色),RNA标签将RNA(红色)结合到树脂支持物上,加入细胞提取物中的蛋白质与RNA结合。洗涤步骤后,Cys4和S1适体方法分别用咪唑和生物素洗脱RNA复合物。底部图,蛋白质微阵列的示意图。体外转录RNA并带上Cy5标记。然后将RNA加入到包含大约9400种蛋白质点样的人蛋白芯片中。洗涤步骤后,使用荧光检测和定量结合在微阵列斑点蛋白的RNA。
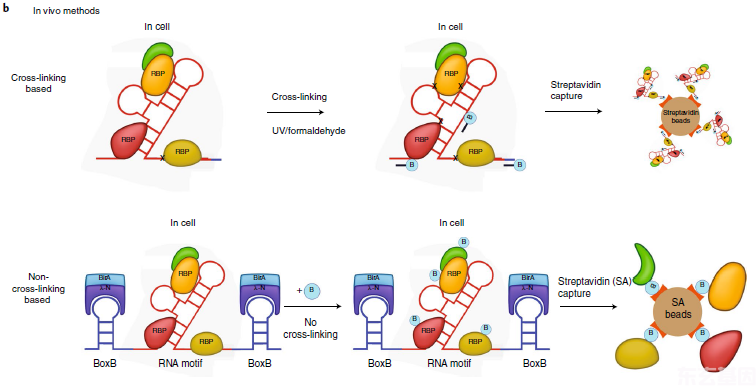
图2 顶部图,体内交联方法的示意图
基于交联的方法使用UV(RAP,PAIR,MS2-BioTRAP,TRIP法)或甲醛交联(CHART,ChIRP法)。生物素化的寡核苷酸探针与感兴趣的RNA杂交,最终纯化RNA和交联蛋白以用于下游分析。底部图,体内非交联方法(RaPID法)的示意图。BoxB RNA茎环(蓝色)位于感兴趣的RNA序列(红色)的侧翼。RaPID(LN-HA-BirA *)融合蛋白与BoxB位点结合,导致生长在含有生物素的培养基中的活细胞中与插入RNA序列相关的蛋白发生生物素酰化。链霉亲和素珠子用于捕获生物素化的蛋白。
2. 体内交联法
研究人员可通过RNA-蛋白质交联鉴定体内两者的相互作用,原理是在变性情况下,排除非共价结合的相互作用,纯化RNA,接着只提取发生交联的蛋白以用于鉴定。
甲醛是一种小的双功能交联剂,可以很容易地渗透细胞并交联2Å距离内的大分子,包括蛋白质—蛋白质,蛋白质—DNA和蛋白质—RNA复合物,形成可逆的共价连接,导致很难区分蛋白质—蛋白质,蛋白质—RNA交联情况下的蛋白。甲醛更倾向于促进亲核赖氨酸残基发生交联。ChIRP和CHART法的原理都是基于甲醛交联法,研究RNA与蛋白的相互作用。
UV光则以零距离和不可逆的共价键形式促进蛋白质与核酸发生交联,有轻度的尿苷交联倾向性,而不会促进蛋白质—蛋白质交联,这点比甲醛交联法更特异,但对于双链RNA的交联效果偏差,对于蛋白的交联效果取决于氨基酸。RAP、PAIR、MS2-BioTRAP和TRIP法的原理都是基于UV交联,但是具体的实验步骤不一样。
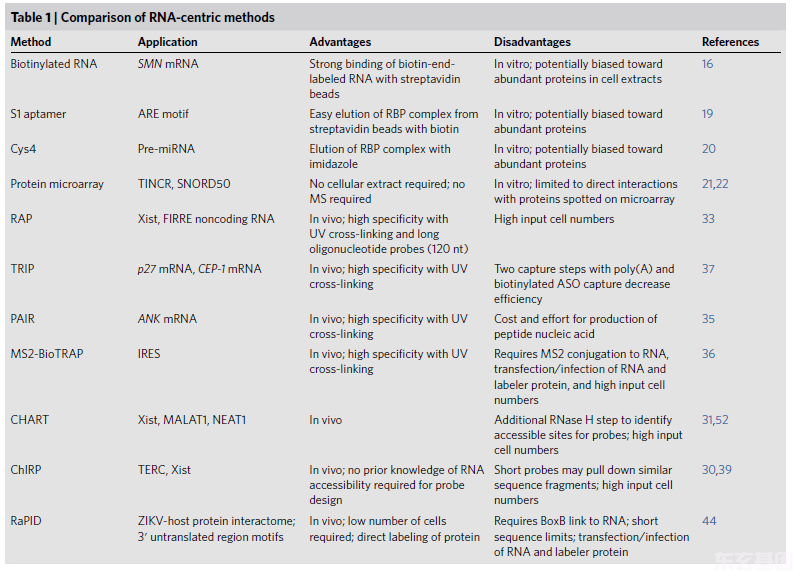
表1 各种RNA-centric法的优点、缺点和应用范围
3. 体内非交联法
近来,不要分子交联研究内源蛋白相互作用的技术,邻近蛋白质组学(Proximity proteomics)也被用于活细胞内以RNA为研究中心的RNA-蛋白质相互作用的研究。生物素连接酶‘Promiscuous’可将生物素转化为活性的biotin-5-AMP,其可共价标记附近的赖氨酸残基,基于这一特性,Promiscuous被用于蛋白-蛋白相互作用的研究。胞内环境是处于还原性状态的,biotin-5-AMP被释放后在20nm距离内会发生淬灭而变成失活的。所以相比于其他蛋白,20nm距离内的蛋白会优先被标记上生物素。RaPID方法用于检测RNA–蛋白相互作用,其原理在于利用这种空间检测限制来检测RBPs,通过用BoxB适体标记感兴趣的RNA,招募λ-N和‘Promiscuous’生物素连接酶的融合蛋白。生物素“传播者”通过λ-N域结合BoxB适体,标记与RNA结合的蛋白。
蛋白组学分析
对于大多数RNA-centric方法的后续分析,一般采取定量质谱MS来挖掘新的RBPs。定量质谱技术又分为标记和无标记质谱法两种 。质谱标记分析法包括SILAC 和iTRAQ,适用于甲醛交联法和体外RNA-centric法,但是这些分析方法的特异性较差,其结果容易被非特异结合的蛋白所干扰,技术要求高,耗费昂贵。无标记质谱分析法的局限性在于无法区分和RNA真正结合的蛋白与非特异结合的蛋白。SAINT分析工具用于分析非定量质谱来源的光谱计数数据,有效地评分RNA-蛋白的真正相互作用的可能性。建议设置两个以上的重复样本,提高说服力和避免非定量质谱分析中出现的假阳性。
如何选择合适的RNA-centric方法
通过上面对各种RNA-centric方法的介绍,那么如何选择最优的研究方案应用于特定的研究问题?主要考虑到三个因素:
1. 首先决定选择体内还是体外方法来研究。一般来说,体外RNA-centric方法用于验证已知的RNA-蛋白的相互结合,而体内RNA-centric方法用于发现新的RNA-蛋白的相互结合;
2. 第二个考虑的因素是RNA丰度。细胞内的RNA丰度越高,用于体内研究RNA-蛋白的相互结合所需的细胞数量越少;
3. 第三个考虑的因素是研究的RNA-蛋白相互作用的强度,主要影响到交联研究方法的选择。
蛋白-centric方法:以感兴趣的蛋白为中心,寻找与该蛋白相互作用的RNAs
蛋白-centric方法的原理,要么直接纯化蛋白,在寻找与之相关的RNAs;要么在某种程度上取决于RNA与感兴趣的蛋白质相关,使用选择性化学修饰的RNA。最常见的研究方法是利用大约254nm UV光照射后,体内蛋白可以化学性地交联核酸,接着纯化感兴趣的蛋白,高通量测序鉴定与之结合的RNAs,这种方法也叫交联免疫沉淀法—高通量测序(CLIP-seq)。但是这种方法局限性在于无法免疫纯化到足够的交联RNA。
1.重点介绍了不同CLIP-seq实验流程,从免疫沉淀纯化蛋白与RNA复合物到PCR扩增检测的过程。
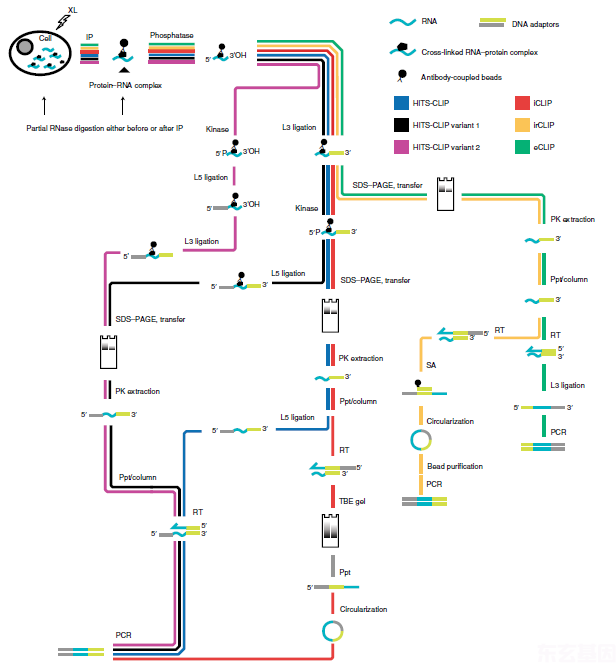
CLIP实验方案图,从免疫沉淀到PCR。重点介绍了代表性的不同类型CLIP-seq的实验步骤。并非所有步骤都包含在内。XL,UV交联; IP,免疫纯化; phosphatase,去除3'磷酸基团; kinase,用于添加5'磷酸; RT,逆转录; L3, RNA或DNA3’端连接适体; L5,5'端适体连接; PK提取,蛋白酶K从硝酸纤维素膜中提取;Ppt /柱,乙醇沉淀或过柱清除核酸; TBE,Tris-borate-EDTA; SA,链霉亲和素。
2. 基于CLIP-seq原理研究与感兴趣蛋白结合的RNAs的不同研究方法的具体优点和缺点
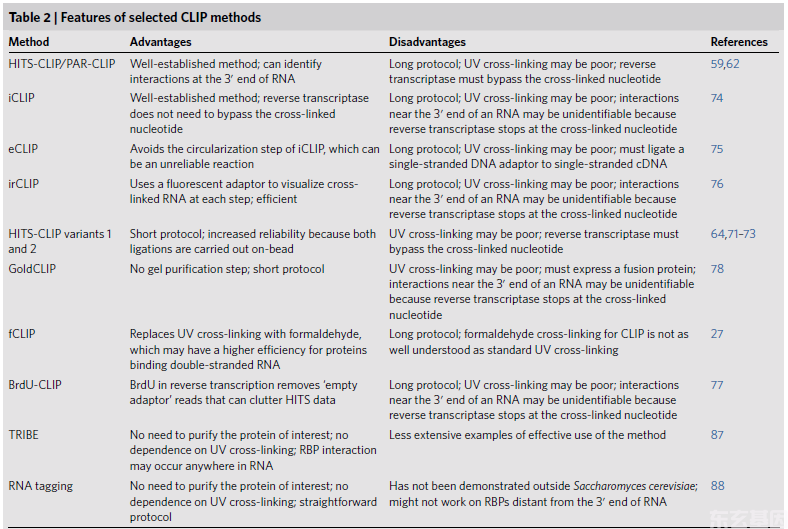
表2 鉴定与感兴趣蛋白结合的RNA的研究方法的优点和缺点(前8种方法基于交联后免疫沉淀的原理,而最后两种方法基于非蛋白纯化,而是依赖于RNA的两种不同化学修饰的原理,鉴定与RBP结合的RNAs)
3. CLIP-seq数据的分析方法
目前还没有通用标准用于CLIP-seq数据分析,这主要取决于研究目的和背景。分析过程中最大的问题在于对CLIP-seq数据的定量。

表3 用于分析CLIP-seq数据的统计方法